Umwelt
Klimawandel trifft Gesundheitssystem
Circular Food Packaging Solutions for the Household – Made from Recycled Materials and Designed for Recycling
Messung von Luftschadstoffen in Echtzeit: ERC Proof of Concept Grant für TU Graz-Physikerin
Eine Lösung für das Energie-Trilemma: Wie sicher ist unsere Energieversorgung?
Warum unser Gehirn nach Linien sucht
Leben
Wissenswertes
PM:Zentrum für Geschlechtersensible Forschung gegründet
ERC Grants: Zwei Forschungsprojekte der LMU ausgezeichnet
Resilienz als Erfolgsfaktor im Tourismus
Deutsche Krebshilfe fordert wissenschaftliche Evaluation von weiteren Krebsfrüherkennungsuntersuchungen
Startup Factory UNITE: Wo Wissenschaft zu Wirtschaft wird / Bundesministerium für Wirtschaft und Energie (BMWE) prämiert UNITE als eines von zehn Leuchtturmprojekten für wissenschaftsbasierte Gründungen
Klima
Nicht immer nur Englisch: Fachartikel in anderen Sprachen helfen, die Artenvielfalt zu bewahren
Ökologische Nachhaltigkeit kommt beim Lausitzer Strukturwandel zu kurz
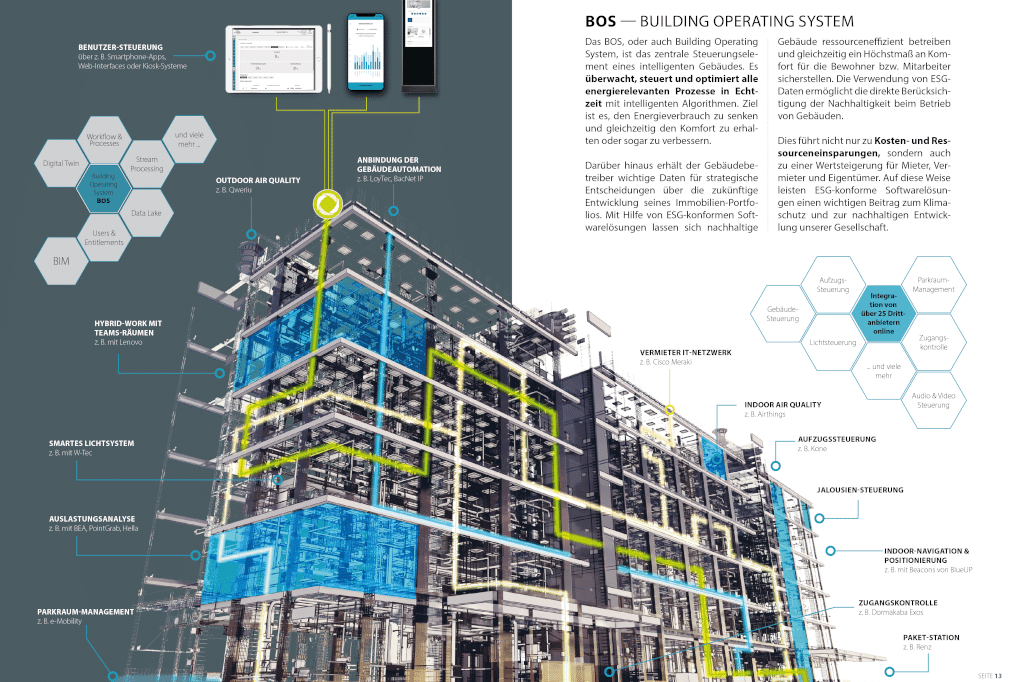
Moderne Collaboration durch digitale Transformation
Aktuell
Klimawandel trifft Gesundheitssystem
Wie die FAU mit AdaptNet Arztpraxen fit für die Zukunft macht Quelle: IDW
Circular Food Packaging Solutions for the Household – Made from Recycled Materials and Designed for Recycling
Das Streben nach einer Kreislaufwirtschaft spielt in der Food- und Verpackungsindustrie eine zentrale Rolle. Daraus resultiert auch die zentrale Fragestellung, wie Lebensmittelverpackungen für den Haushalt…
Messung von Luftschadstoffen in Echtzeit: ERC Proof of Concept Grant für TU Graz-Physikerin
Für die Entwicklung eines tragbaren Geräts, das in Sekundenbruchteilen mehrere Schadstoffe zugleich misst, erhält Birgitta Schultze-Bernhardt eine Förderung des Europäischen Forschungsrats. Quelle: IDW
Eine Lösung für das Energie-Trilemma: Wie sicher ist unsere Energieversorgung?
Wir müssen unser Energiesystem nachhaltiger gestalten – aber welcher Weg dazu ist der beste? Um die Energieversorgung der Zukunft planen zu können, brauchen Entscheidungsträgerinnen und…
PM:Zentrum für Geschlechtersensible Forschung gegründet
Eingebunden lt. EuGH – Beschluss vom 21.10.2014 – Az. C-348/13
Drei ERC Proof of Concept Grants für Göttinger Wissenschaftler
Der Göttinger Chemiker Prof. Dr. Lutz Ackermann und die Göttinger Biophysiker Prof. Dr. Timo Betz und Prof. Dr. Jörg Enderlein haben jeweils einen Proof of…
Kryo-Elektronenmikroskopie – Reaktionszyklus eines Enzyms zur CO2-Fixierung entschlüsselt
Hochauflösende Kryo-Elektronenmikroskopie macht es möglich, komplexe enzymatische Prozesse detailliert zu untersuchen. Einem Forschungsteam der Universität Potsdam und der Humboldt-Universität zu Berlin ist es mit dieser…
ERC Grants: Zwei Forschungsprojekte der LMU ausgezeichnet
Eingebunden lt. EuGH – Beschluss vom 21.10.2014 – Az. C-348/13
Resilienz als Erfolgsfaktor im Tourismus
Eingebunden lt. EuGH – Beschluss vom 21.10.2014 – Az. C-348/13
Deutsche Krebshilfe fordert wissenschaftliche Evaluation von weiteren Krebsfrüherkennungsuntersuchungen
Eingebunden lt. EuGH – Beschluss vom 21.10.2014 – Az. C-348/13
Neues Tool SNAP-tag2 markiert Proteine schneller und intensiver
Das Protein SNAP-tag ist ein leistungsstarkes Werkzeug, um Proteine mit synthetischen Fluorophoren für die Biobildgebung zu markieren. Wissenschaftler*innen des Max-Planck-Instituts für medizinische Forschung in Heidelberg…
Schweizer Virusgenom der Spanischen Grippe entschlüsselt
Forschende der Universitäten Basel und Zürich konnten anhand eines historischen Präparats der medizinischen Sammlung der UZH erstmals das Virusgenom der Grippepandemie von 1918-1920 in der…